n <- 100
set.seed(48967365)
X <- sample(c("A","B","C"),100,replace=TRUE)
Y <- rep(0,n)
set.seed(487365)
Y[X=="A"] <- rbinom(sum(X=="A"),size=1,prob=0.95)
set.seed(4878365)
Y[X=="B"] <- rbinom(sum(X=="B"),size=1,prob=0.95)
set.seed(4653965)
Y[X=="C"] <- rbinom(sum(X=="C"),size=1,prob=0.05)
Y <- factor(Y)
donnees<-data.frame(Y,X)11 Régression logistique
Exercice 1 (Questions de cours)
- A
- A
- B
- A
- A
- A
- B
- A
Exercice 2 (Interprétation des coefficients)
On génère l’échantillon.
On ajuste le modèle avec les contraintes par défaut.
model1 <- glm(Y~X,data=donnees,family=binomial) summary(model1)Call: glm(formula = Y ~ X, family = binomial, data = donnees) Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) 2.2336 0.6075 3.677 0.000236 *** XB 0.6568 0.9470 0.694 0.487977 XC -5.6348 1.1842 -4.758 1.95e-06 *** --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 129.489 on 99 degrees of freedom Residual deviance: 44.218 on 97 degrees of freedom AIC: 50.218 Number of Fisher Scoring iterations: 6On obtient les résultats du test de Wald sur la nullité des paramètres \(\beta_0,\beta_2\) et \(\beta_3\).
On change la modalité de référence.
model2 <- glm(Y~C(X,base=3),data=donnees,family=binomial) summary(model2)Call: glm(formula = Y ~ C(X, base = 3), family = binomial, data = donnees) Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) -3.401 1.017 -3.346 0.00082 *** C(X, base = 3)A 5.635 1.184 4.758 1.95e-06 *** C(X, base = 3)B 6.292 1.249 5.035 4.77e-07 *** --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 129.489 on 99 degrees of freedom Residual deviance: 44.218 on 97 degrees of freedom AIC: 50.218 Number of Fisher Scoring iterations: 6On obtient les résultats du test de Wald sur la nullité des paramètres \(\beta_0,\beta_1\) et \(\beta_2\).
On remarque que dans model1 on accepte la nullité de \(\beta_2\) alors qu’on la rejette dans model2. Ceci est logique dans la mesure où ces tests dépendent de la contrainte identifiante choisie. Dans model1 le test de nullité de \(\beta_2\) permet de vérifier si \(B\) à un effet similaire à \(A\) sur \(Y\). Dans model2, on compare l’effet de \(B\) à celui de \(C\). On peut donc conclure \(A\) et \(B\) ont des effets proches sur \(Y\) alors que \(B\) et \(C\) ont un impact différent. Ceci est logique vu la façon dont les données ont été générées.
Tester l’effet global de \(X\) sur \(Y\) revient à tester si les coefficients \(\beta_1,\beta_2\) et \(\beta_3\) sont égaux, ce qui, compte tenu des contraintes revient à considérer les hypothèses nulles :
- \(\beta_2=\beta_3=0\) dans model1 ;
- \(\beta_1=\beta_2=0\) dans model2.
On peut effectuer les tests de Wald ou du rapport de vraisemblance. On obtient les résultats du rapport de vraisemblance avec :
library(car) Anova(model1,type=3,test.statistic="LR")Analysis of Deviance Table (Type III tests) Response: Y LR Chisq Df Pr(>Chisq) X 85.271 2 < 2.2e-16 *** --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Anova(model2,type=3,test.statistic="LR")Analysis of Deviance Table (Type III tests) Response: Y LR Chisq Df Pr(>Chisq) C(X, base = 3) 85.271 2 < 2.2e-16 *** --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1On remarque ici que ces deux tests sont identiques : ils ne dépendent pas de la contrainte identifiante choisie.
Exercice 3 (Séparabilité)
On génère l’échantillon demandé.
set.seed(1234) X <- c(runif(50,-1,0),runif(50,0,1)) set.seed(5678) Y <- c(rep(0,50),rep(1,50)) df <- data.frame(X,Y)Le graphe s’obtient avec :
beta <- seq(0,100,by=0.01) log_vrais <- function(X,Y,beta){ LV <- rep(0,length(beta)) for (i in 1:length(beta)){ Pbeta <- exp(beta[i]*X)/(1+exp(beta[i]*X)) LV[i] <- sum(Y*X*beta[i]-log(1+exp(X*beta[i]))) # gradln[i] <- t(Xb)%*%(Yb-Pbeta) } return(LV) } LL <- log_vrais(X,Y,beta) plot(beta,LL,type="l")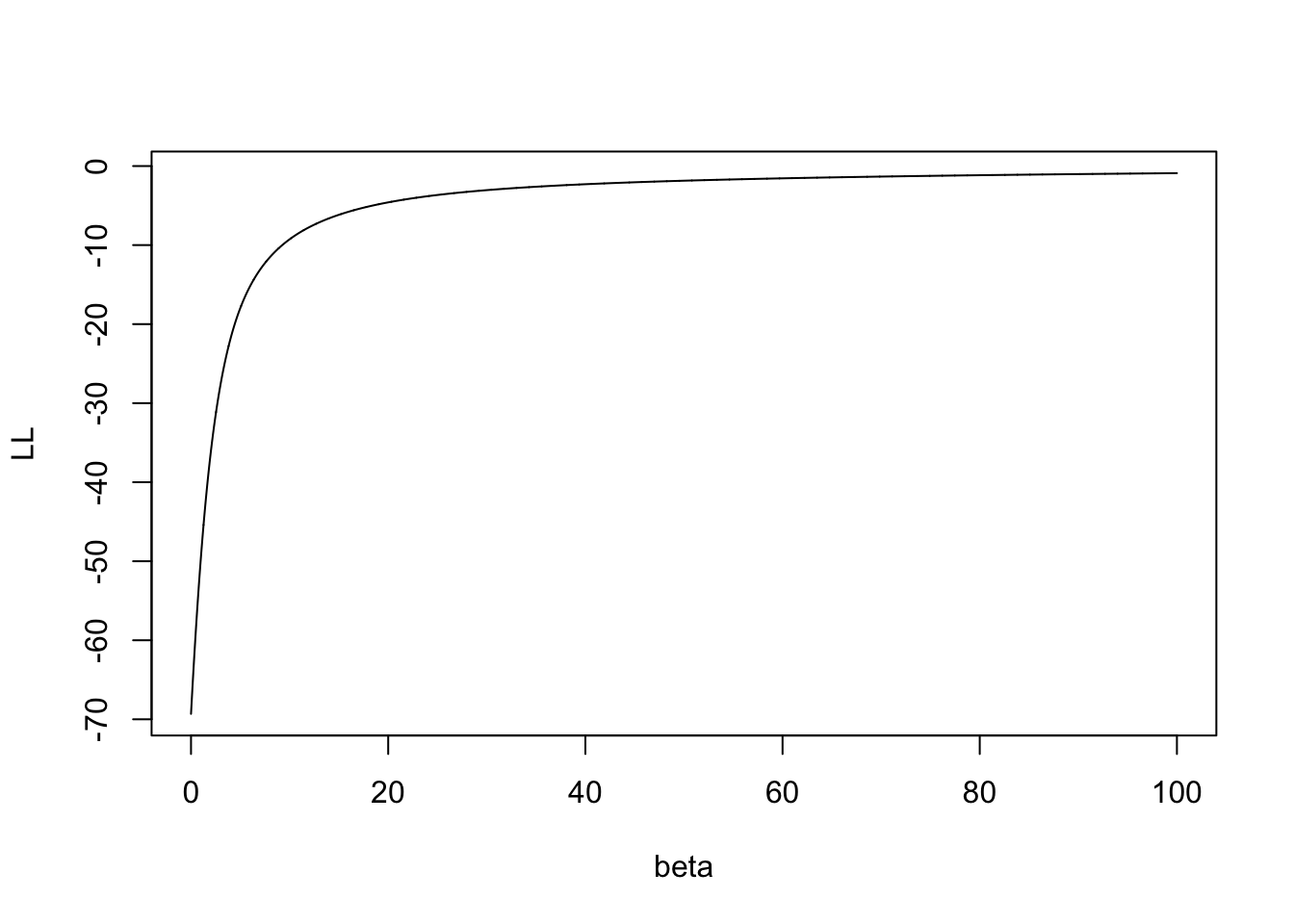
On obtient un avertissement qui nous dit que l’algorithme d’optimisation n’a pas convergé.
model <- glm(Y~X-1,data=df,family="binomial") model$coefX 1999.371Le changement proposé supprime la séparabilité des données. On obtient bien un maximum fini pour cette nouvelle vraisemblance.
Y1 <- Y;Y1[1] <- 1 LL1 <- log_vrais(X,Y1,beta) plot(beta,LL1,type="l")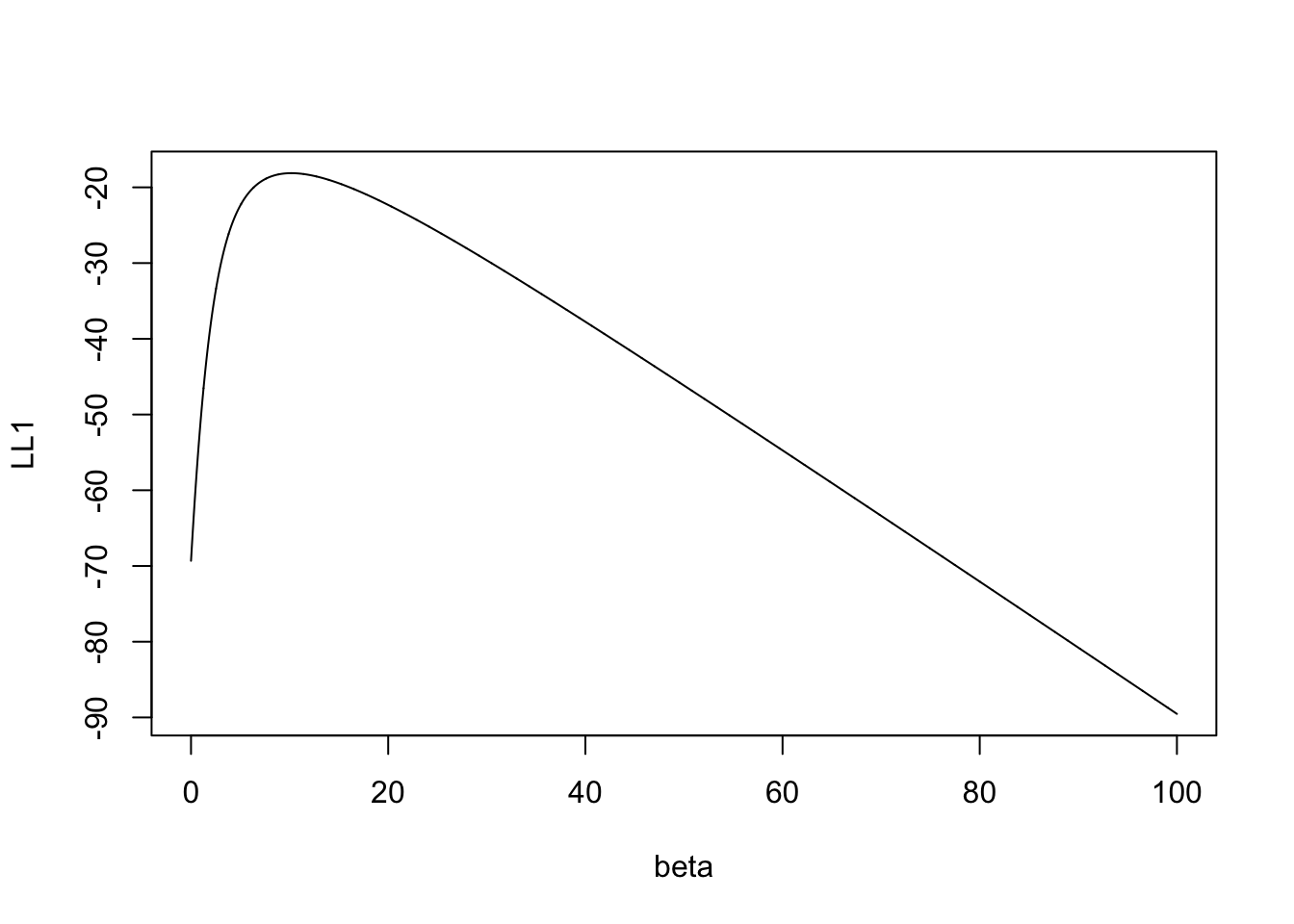
model1 <- glm(Y1~X-1,family="binomial") model1$coefX 10.17868
Exercice 4 (Matrice hessienne) Le gradient de la log-vraisemblance en \(\beta\) est donné par \(\nabla \mathcal L(Y,\beta)=X'(Y-P_\beta)\). Sa \(j\)ème composante vaut \[\frac{\partial\mathcal L}{\partial\beta_j}(\beta)=\sum_{i=1}^nx_{ij}(y_i-p_\beta(x_i)).\]
On peut donc calculer la drivée par rapport à \(\beta_\ell\) : \[\begin{align*} \frac{\partial\mathcal L}{\partial\beta_j\partial\beta_\ell}(\beta)= & \frac{\partial}{\partial\beta_\ell}\left[ \sum_{i=1}^nx_{ij}\left(y_i-\frac{\exp(x_i'\beta)}{1+\exp(x_i'\beta)}\right)\right] \\ =& -\sum_{i=1}^nx_{ij}x_{i\ell}\frac{\exp(x_i'\beta)}{[1+\exp(x_i'\beta)]^2} \\ =& -\sum_{i=1}^nx_{ij}x_{i\ell}p_\beta(x_i)(1-p_\beta(x_i)). \end{align*}\] Matriciellement on déduit donc que la hessienne vaut \[\nabla^2\mathcal L(Y,\beta)=-X'W_\beta X,\] où \(W_\beta\) est la matrice \(n\times n\) diagonale dont le \(i\)ème terme de la diagonale vaut \(p_\beta(x_i)(1-p_\beta(x_i))\). Par ailleurs, comme pour tout \(i=1,\dots,n\), on a \(p_\beta(x_i)(1-p_\beta(x_i))>0\) et que \(X\) est de plein rang, on déduit que \(X'W_\beta X\) est définie positive et par conséquent que la hessienne est définie négative.
Exercice 5 (Modèles avec R) On importe les données
panne <- read.table("../donnees/panne.txt",header=T)
head(panne) etat age marque
1 0 4 A
2 0 2 C
3 0 3 C
4 0 9 B
5 0 7 B
6 0 6 ALa commande
model <- glm(etat~.,data=panne,family=binomial) modelCall: glm(formula = etat ~ ., family = binomial, data = panne) Coefficients: (Intercept) age marqueB marqueC 0.47808 0.01388 -0.41941 -1.45608 Degrees of Freedom: 32 Total (i.e. Null); 29 Residual Null Deviance: 45.72 Residual Deviance: 43.5 AIC: 51.5ajuste le modèle \[\log\left(\frac{p_\beta(x)}{1-p_\beta(x)}\right)=\beta_0+\beta_1x_1+\beta_2\mathsf{1}_{x_2=B}+\beta_3\mathsf{1}_{x_2=C}\] où \(x_1\) et \(x_2\) désigne respectivement les variables age et marque. On obtient les estimateurs avec
coef(model)(Intercept) age marqueB marqueC 0.47808311 0.01388395 -0.41941071 -1.45608147Il s’agit des tests de Wald pour tester l’effet des variables dans le modèle. Pour l’effet de marque, on va par exemple tester \[H_0:\beta_2=\beta_3=0\quad\text{contre}\quad H_1:\beta_2\neq 0\text{ ou }\beta_3\neq 0.\] Sous \(H_0\) la statistique de Wald suit une loi du \(\chi^2\) à 4-2=2 degrés de liberté. Pour le test de la variable age le nombre de degrés de liberté manquant est 1. On retrouve cela dans la sortie
library(car) Anova(model,type=3,test.statistic="Wald")Analysis of Deviance Table (Type III tests) Response: etat Df Chisq Pr(>Chisq) (Intercept) 1 0.3294 0.5660 age 1 0.0218 0.8826 marque 2 1.9307 0.3809Il s’agit cette fois du test du rapport de vraisemblance. Les degrés de liberté manquants sont identiques.
Anova(model,type=3,test.statistic="LR")Analysis of Deviance Table (Type III tests) Response: etat LR Chisq Df Pr(>Chisq) age 0.02189 1 0.8824 marque 2.09562 2 0.3507Le modèle s’écrit \[\log\left(\frac{p_\beta(x)}{1-p_\beta(x)}\right)=\beta_0+\beta_1\mathsf{1}_{x_2=A}+\beta_2\mathsf{1}_{x_2=B}.\]
Le modèle ajusté ici est \[\log\left(\frac{p_\beta(x)}{1-p_\beta(x)}\right)=\gamma_0+\gamma_1\mathsf{1}_{x_2=B}+\gamma_2\mathsf{1}_{x_2=C}.\] Par identification on a \[\begin{cases} \beta_0+\beta_1=\gamma_0 \\ \beta_0+\beta_2=\gamma_0+\gamma_1 \\ \beta_0=\gamma_0+\gamma_2 \\ \end{cases} \Longleftrightarrow \begin{cases} \beta_0=\gamma_0+\gamma_2 \\ \beta_1=-\gamma_2 \\ \beta_2=\gamma_1-\gamma_2 \\ \end{cases} \Longrightarrow \begin{cases} \widehat\beta_0=-0.92 \\ \widehat\beta_1=1.48 \\ \widehat\beta_2=1.05 \\ \end{cases}\] On peut retrouver ces résultats avec
glm(etat~C(marque,base=3),data=panne,family="binomial")Call: glm(formula = etat ~ C(marque, base = 3), family = "binomial", data = panne) Coefficients: (Intercept) C(marque, base = 3)A C(marque, base = 3)B -0.9163 1.4759 1.0498 Degrees of Freedom: 32 Total (i.e. Null); 30 Residual Null Deviance: 45.72 Residual Deviance: 43.52 AIC: 49.52
Il y a interaction si l’age agit différemment sur la panne en fonction de la marque.
Le modèle ajusté sur R est \[\log\left(\frac{p_\beta(x)}{1-p_\beta(x)}\right)=\delta_0+\delta_1\mathsf{1}_{x_2=B}+\delta_2\mathsf{1}_{x_2=C}+\delta_3x_1+\delta_4x_1\mathsf{1}_{x_2=B}+\delta_5x_1\mathsf{1}_{x_2=C}.\] On obtient ainsi par identification : \[\begin{cases} \alpha_0=\delta_0\\ \alpha_1=\delta_3\\ \beta_0=\delta_0+\delta_1\\ \beta_1=\delta_3+\delta_4\\ \gamma_0=\delta_0+\delta_2\\ \gamma_1=\delta_3+\delta_5 \end{cases}\] On peut ainsi en déduire les valeurs des estimateurs que l’on peut retrouver avec la commande :
glm(etat~-1+marque+marque:age,data=panne,family="binomial")Call: glm(formula = etat ~ -1 + marque + marque:age, family = "binomial", data = panne) Coefficients: marqueA marqueB marqueC marqueA:age marqueB:age marqueC:age 0.23512 0.43375 -2.19633 0.05641 -0.05547 0.27228 Degrees of Freedom: 33 Total (i.e. Null); 27 Residual Null Deviance: 45.75 Residual Deviance: 42.62 AIC: 54.62
Exercice 6 (Interprétation)
df <- read.csv("../donnees/logit_ex6.csv")
mod <- glm(Y~.,data=df,family=binomial)
mod1 <- glm(Y~X1,data=df,family=binomial)
summary(mod)
Call:
glm(formula = Y ~ ., family = binomial, data = df)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -0.57866 0.11926 -4.852 1.22e-06 ***
X1 -0.19471 0.06556 -2.970 0.00298 **
X2 0.31899 0.04404 7.244 4.36e-13 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 693.15 on 499 degrees of freedom
Residual deviance: 618.26 on 497 degrees of freedom
AIC: 624.26
Number of Fisher Scoring iterations: 4summary(mod1)
Call:
glm(formula = Y ~ X1, family = binomial, data = df)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.001092 0.089499 0.012 0.990
X1 -0.020467 0.051733 -0.396 0.692
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 693.15 on 499 degrees of freedom
Residual deviance: 692.99 on 498 degrees of freedom
AIC: 696.99
Number of Fisher Scoring iterations: 3On remarque que la nullité du paramètre associé à X1 est accepté dans le modèle avec uniquement X1 alors qu’elle est refusée lorsqu’on considère X1 et X2 dans le modèle.
Exercice 7 (Tests à la main)
Le modèle s’écrit \[log\left(\frac{p_\beta(x)}{1-p_\beta(x)}\right)=\beta_0+\beta_1x.\]
La log vraisemblance s’obtient avec
p <- c(0.76,0.4,0.6,0.89,0.35) Y <- c(1,0,0,1,1) L1 <- log(prod(p^Y*(1-p)^(1-Y))) L1[1] -2.867909On calcule les écart-type des estimateurs
X1 <- c(0.47,-0.55,-0.01,1.07,-0.71) X <- matrix(c(rep(1,5),X1),ncol=2) W <- diag(p*(1-p)) SIG <- solve(t(X)%*%W%*%X) sig <- sqrt(diag(SIG)) sig[1] 1.023252 1.744935On en déduit les statistiques de test :
beta <- c(0.4383,1.5063) beta/sig[1] 0.4283401 0.8632411On peut faire le test de Wald et du rapport de vraisemblance.
La statistique de test vaut 0.8632411, on obtient donc la probabilité critique
2*(1-pnorm(0.8632411))[1] 0.3880049On peut également effectuer un test du rapport de vraisemblance. Le modèle null sans X1 a pour log-vraisemblance
p0 <- 3/5 L0 <- log(prod(p0^Y*(1-p0)^(1-Y))) L0[1] -3.365058La statistique de test vaut donc
2*(L1-L0)[1] 0.9942984et la probabilité critique vaut
1-pchisq(2*(L1-L0),df=1)[1] 0.3186941On peut retrouver (aux arrondis près) les résultats de l’exercice avec
X <- c(0.47,-0.55,-0.01,1.07,-0.71) Y <- c(1,0,0,1,1) df <- data.frame(X,Y) model <- glm(Y~X,data=df,family="binomial") logLik(model)'log Lik.' -2.898765 (df=2)Anova(model,type=3,test.statistic = "Wald")Analysis of Deviance Table (Type III tests) Response: Y Df Chisq Pr(>Chisq) (Intercept) 1 0.1846 0.6675 X 1 0.7552 0.3848Anova(model,type=3,test.statistic = "LR")Analysis of Deviance Table (Type III tests) Response: Y LR Chisq Df Pr(>Chisq) X 0.93259 1 0.3342
Exercice 8 (Vraisemblance du modèle saturé)
Les variables \((y_t,t=1,\dots,y_T)\) étant indépendantes et de loi binomiales \(B(n_t,p_t)\), la log-vraisemblance est donnée par \[\begin{align*} \mathcal L_{\text{sat}}(Y,p)= & \log\left(\prod_{t=1}^T \begin{pmatrix} n_t\\ \tilde y_t \end{pmatrix} p_t^{\tilde y_t}(1-p_t)^{n_t-\tilde y_t}\right) \\ = & \sum_{t=1}^T\left(\log \begin{pmatrix} n_t\\ \tilde y_t \end{pmatrix} +\tilde y_t\log(p_t)+(n_t-\tilde y_t)\log(1-p_t)\right) \end{align*}\]
La dérivée de la log-vraisemblance par rapport à \(p_t\) s’écrit \[\frac{\tilde y_t}{p_t}-\frac{n_t-\tilde y_t}{1-p_t}.\] Cette dérivée s’annule pour \[\widehat p_t=\frac{\tilde y_t}{n_t}.\]
On note \(\widehat \beta\) l’EMV du modèle logistique et \(p_{\widehat\beta}\) le vecteur qui contient les valeurs ajustées \(p_{\widehat\beta}(x_t),t=1,\dots,T\). On a pour tout \(\beta\in\mathbb R^p\) : \[\mathcal L(Y,\beta)\leq\mathcal L(Y,\widehat\beta)=\mathcal L_{\text{sat}}(Y,p_{\widehat\beta})\leq L_{\text{sat}}(Y,\widehat p_t).\]
Exercice 9 (Intervalle de confiance profilé)
artere <- read.table("../donnees/artere.txt",header=T) modele <- glm(chd~age,data=artere,family=binomial) B0 <- coef(modele) OriginalDeviance <- modele$deviancealpha <- 0.05stderr <- summary(modele)$coefficients[, "Std. Error"] delta <- sqrt(qchisq((1-alpha/4),df=1))* stderr[2] /5 grille <- B0[2]+(-10):10*deltaOn a \[\begin{align*} \mathcal D_1&=-2(\mathcal L(Y,\hat\beta)-\mathcal L_{sat}) \end{align*}\] Pour celle avec l’offset \(K_i=x_i\beta_2^*\) elle vaut \[\begin{align*} \mathcal D_o&=-2(\mathcal L(Y,K,\hat\beta_1)-\mathcal L_{sat}) \end{align*}\] où \(\hat \beta_1\) maximise \(\mathcal L(Y,K,\hat\beta_1)\) c’est à dire \(\mathcal L(Y,K,\hat\beta_1)=l(\beta_2^*)\) et nous avons donc \[\begin{align*} \mathcal D_o - \mathcal D_1= 2(\mathcal L(Y,\hat\beta)-\mathcal L(Y,K,\beta_1)= 2(\mathcal L(Y,\hat\beta)-l(\beta_2^*))=P(\beta_2^*). \end{align*}\]
profil2 <- rep(0,length(grille)) for (k in 1:length(grille)) { modeleo <- glm(chd~1,family=binomial,offset=artere[,"age"]*grille[k],data=artere) profil2[k] <- modeleo$deviance - OriginalDeviance }profil <- sign(-10:10)*sqrt(profil2)spline(x=profil,y=grille,xout=c(-sqrt(qchisq(1-alpha,1)),sqrt(qchisq(1-alpha,1))))$y[1] 0.0669275 0.1620014confint(modele)2.5 % 97.5 % (Intercept) -7.72587162 -3.2461547 age 0.06693158 0.1620067